分子間相互作用に基づくシグナル伝達網解析のための無細胞プロテオーム技術の開発
 研究代表者
研究代表者
澤崎達也
愛媛大学プロテオサイエンスセンター無細胞生命科学部門
http://www.pros.ehime-u.ac.jp/cell-free/
研究概要
細胞内シグナル伝達の多くは、タンパク質同士が相互作用し、翻訳後修飾などを介して下流へと情報が伝えられていく。このシグナル伝達経路を分子レベルで理解するためには、シグナル伝達に関わるタンパク質の相互作用パートナーを見出すことは非常に重要である。これまでの研究においては、培養細胞を用いた免疫沈降や質量分析、さらには酵母2ハイブリッドシステム等による解析により、多くのシグナル伝達に関わる重要な因子が同定されてきた。しかし、これら生細胞を用いた解析では、細胞内において発現量が著しく低いタンパク質や、限られた細胞種でのみ発現するタンパク質を同定することは非常に難しかった。
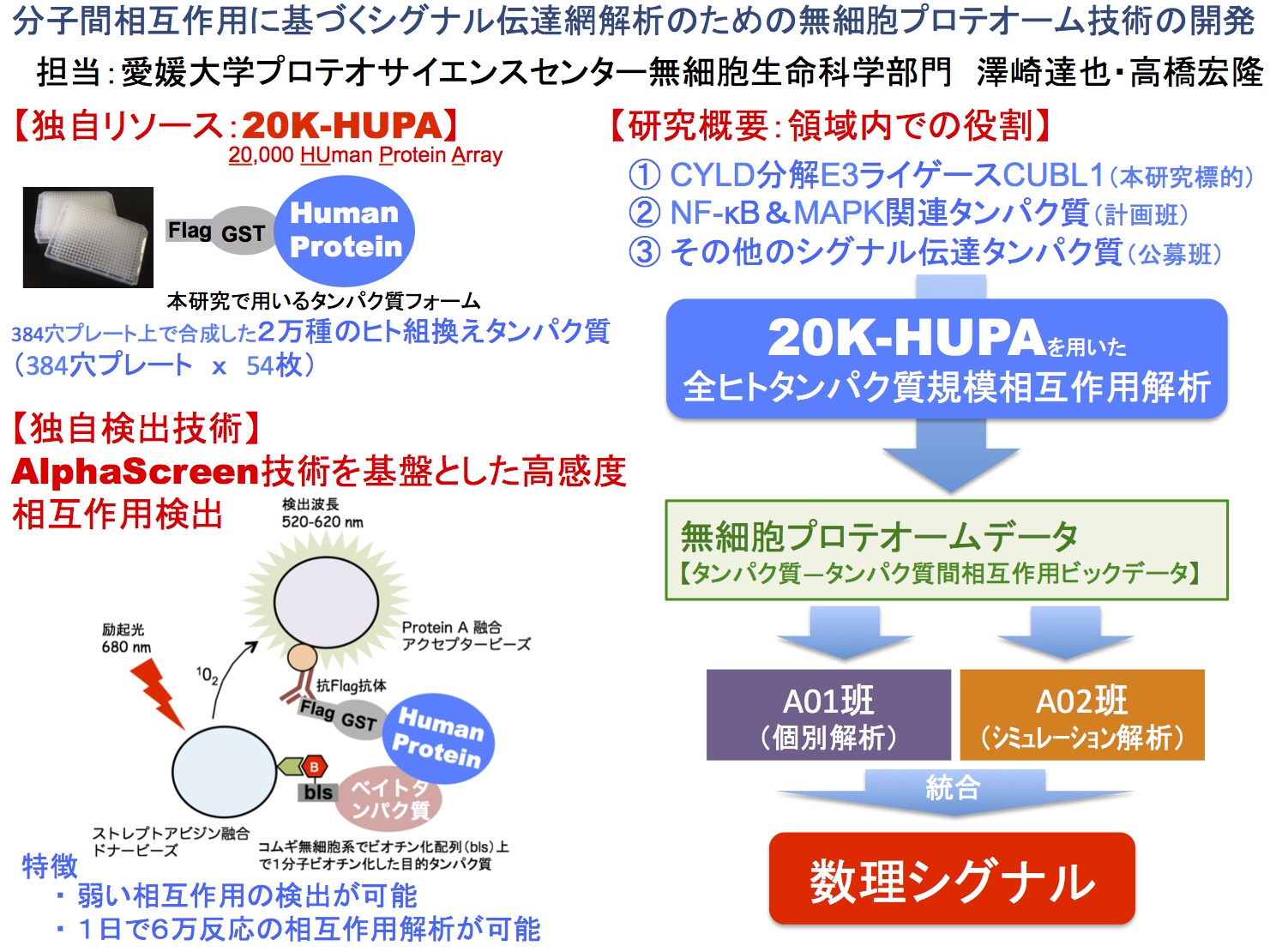
我々は、愛媛大学において独自に開発したコムギ無細胞タンパク質合成系を用いて、ハイスループットな組換えタンパク質の合成および機能解析技術の開発を行ってきた。さらに本技術を応用し、ヒトの約2万種類の遺伝子を鋳型に組換えタンパク質として合成・アレイ化した20K-Human Protein Array(20K-HUPA)と、AlphaScreenを用いた高感度相互作用解析を基盤に、ヒトゲノムにコードされるほぼ全てのタンパク質を対象に、標的タンパク質に相互作用するタンパク質を数時間でスクリーニングできる解析技術の開発に成功した。
本研究では、我々が独自に同定・解析を進めている、脱ユビキチン化酵素CYLDの責任E3リガーゼCUBL1に着目し、20K-HUPAによってCUBL1の相互作用パートナーの同定を目指す。我々の先行研究から、CUBL1はNF-κBの負の制御因子であるCYLDを分解することで、NF-κBシグナルを亢進させることを見出したが、最近になってCUBL1がCYLDの他にRIP-1やLUBACなど、NF-κBシグナルにおいて中心的役割を担う複数のタンパク質と相互作用することが明らかになった。これらの結果から、CUBL1が細胞内において様々なタンパク質と相互作用し、複雑多岐なメカニズムでNF-κBを制御している可能性が強く示唆され、この全容を明らかとすることでNF-κBの新規制御ネットワークの同定を試みる。
さらに本領域においては、計画班や公募班が解析対象としている他のシグナル伝達タンパク質の相互作用パートナーとなるタンパク質を20K-HUPAにより網羅的に同定し、タンパク質間相互作用ビックデータの取得を進める。それらのデータを、種々の個別解析で世界をリードするA01班、およびシミュレーションチームであるA02班に提供することにより、新しい“数理シグナル”解析の方法論構築を目指す。
参考文献
- Yano T, Takeda H, Uematsu A, Yamanaka S, Nomura S, Nemoto K, Iwasaki T, Takahashi H, Sawasaki T. AGIA Tag System Based on a High Affinity Rabbit Monoclonal Antibody against Human Dopamine Receptor D1 for Protein Analysis. PLoS One. 2016 Jun 6;11(6):e0156716. doi: 10.1371/journal.pone.0156716.
- Takahashi H, Uematsu A, Yamanaka S, Imamura M, Nakajima T, Doi K, Yasuoka S, Takahashi C, Takeda H, Sawasaki T. Establishment of a Wheat Cell-Free Synthesized Protein Array Containing 250 Human and Mouse E3 Ubiquitin Ligases to Identify Novel Interaction between E3 Ligases and Substrate Proteins. PLoS One. 2016 Jun 1;11(6):e0156718. doi: 10.1371/journal.pone.0156718.
- Takeda H, Ogasawara T, Ozawa T, Muraguchi A, Jih PJ, Morishita R, Uchigashima M, Watanabe M, Fujimoto T, Iwasaki T, Endo Y, Sawasaki T. Production of monoclonal antibodies against GPCR using cell-free synthesized GPCR antigen and biotinylated liposome-based interaction assay. Sci Rep. 2015 Jun 10;5:11333. doi: 10.1038/srep11333.
- Nemoto K, Takemori N, Seki M, Shinozaki K, Sawasaki T. Members of the Plant CRK-superfamily are Capable of trans-/auto-Phosphorylation of Tyrosine Residues. J Biol Chem. 2015 Jul 3;290(27):16665-77. doi: 10.1074/jbc.M114.617274.
- Iwasaki T, Katayama T, Kohama K, Endo Y, Sawasaki T. Myosin phosphatase is inactivated by caspase-3 cleavage and phosphorylation of myosin phosphatase targeting subunit 1 during apoptosis. Mol Biol Cell. 2013 Mar;24(6):748-56. doi: 10.1091/mbc.E11-08-0740.
- Tadokoro D, Takahama S, Shimizu K, Hayashi S, Endo Y, Sawasaki T. Characterization of a caspase-3-substrate kinome using an N- and C-terminally tagged protein kinase library produced by a cell-free system. Cell Death & Dis. 2010 Oct 28;1:e84. doi: 10.1038/cddis.2010.65.
- Takai K, Sawasaki T, Endo Y. Practical cell-free protein synthesis system using purified wheat embryos. Nat Protoc. 2010;5(2):227-238. doi: 10.1038/nprot.2009.207.
- Sawasaki T, Ogasawara T, Morishita R, Endo Y. A cell-free protein synthesis system for high-throughput proteomics. Proc Natl Acad Sci U S A. 2002 Nov 12;99(23):14652-7.
- Madin K, Sawasaki T, Ogasawara T, Endo Y. A highly efficient and robust cell-free protein synthesis system prepared from wheat embryos: plants apparently contain a suicide system directed at ribosomes. Proc Natl Acad Sci U S A. 2000 Jan 18;97(2):559-64.








