擬似的な一細胞ダイナミクスの再構成による時空間的な遺伝子発現制御機構の解明
 研究代表者
研究代表者
前原一満
九州大学生体防御医学研究所
http://tx.bioreg.kyushu-u.ac.jp
研究概要
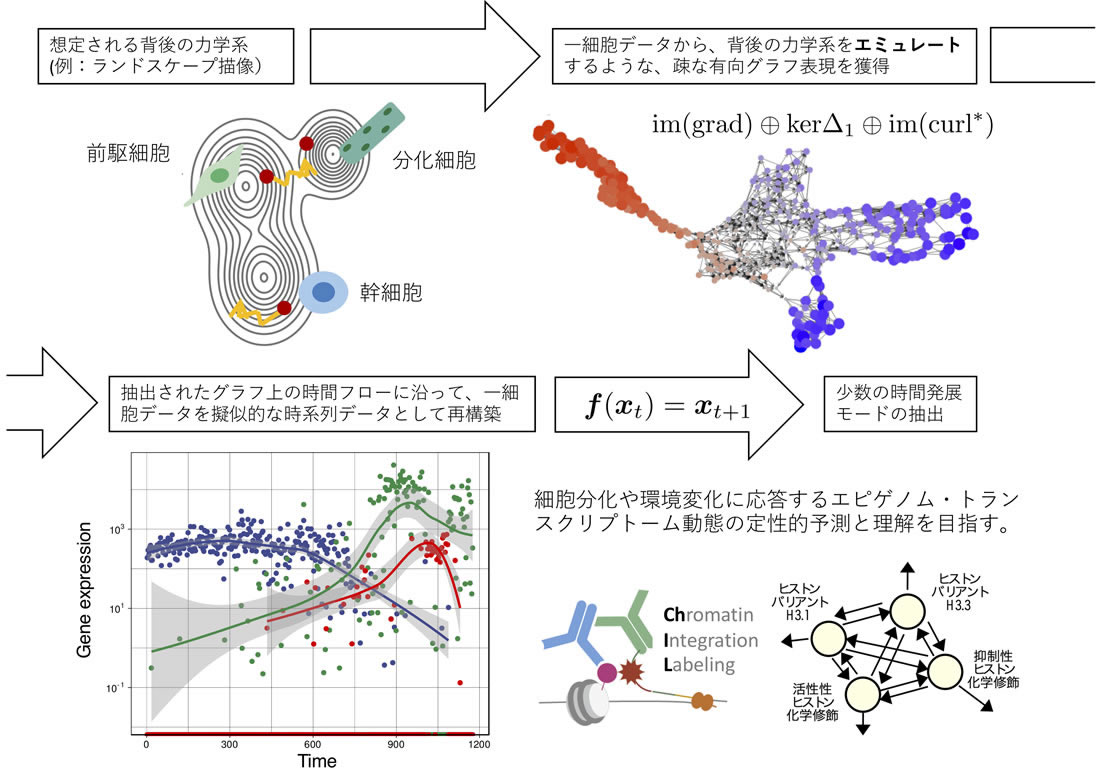
細胞分化における選択的な遺伝情報獲得のメカニズムを理解するため、これまで膨大な数の転写制御因子やそれらが結合するゲノム上の制御配列の組み合わせが数多く同定されてきた。これら多種多様な分子の組み合わせは、ゲノムの構造に大きな自由度をもたらしている。しかしながら、刺激への応答においてゲノムが果たすべき役割は、各遺伝子についての転写活性化、あるいは抑制である。すなわち、大自由度の系でありながら、そのダイナミクスは、必要な応答の種類に限った少数のモードによって表現可能であると考えられる。
近年発展の目覚ましい単一かつ多数細胞を対象とする一細胞計測技術は、とりわけ遺伝子発現解析において一般化しつつある。細胞集団の分類や未知集団の同定においては強力なツールであるが、個々の細胞の時間情報が欠落している現状のままでは、一細胞データから背後のダイナミクスを復元することはできない。そこで本研究では、まず、一細胞トランスクリプトームデータに不足した時間情報をデータ自身から再構築する。時間構造の構築には、現在開発中のホッジ分解による一細胞データの解析手法を用いることを計画している。物理・数学の分野では、ベクトル場の流れを一意に分解できるとするヘルムホルツ-ホッジ分解と呼ばれる定理が知られており、データ点をつなぎ合わせた離散のグラフ構造においても、組合せ論的ホッジ分解を定義できる。直感的な解釈が容易なベクトル解析のアナロジーを持つホッジ分解のフレームワークを一細胞データの解析に応用することで、観測データの背後の力学系をエミュレートする疎なグラフとして再構築することができる。そして、抽出した時間フローに沿って並び替えた一細胞プロファイルを用いて推定した時間発展演算子から、主要なモードを抽出する。本計画によって、分化や組織損傷等の環境変化に応答するエピゲノム・トランスクリプトーム動態の定性的予測や理解を支援する技術基盤の開発を目指す。
参考文献
- Maehara, Kazumitsu, and Yasuyuki Ohkawa. 2019. “Modeling Latent Flows on Single-Cell Data Using the Hodge Decomposition.” BioRxiv. https://doi.org/10.1101/592089.
- Harada, Akihito†, Kazumitsu Maehara†, Tetsuya Handa†, Yasuhiro Arimura, Jumpei Nogami, Yoko Hayashi-Takanaka, Katsuhiko Shirahige, Hitoshi Kurumizaka, Hiroshi Kimura, and Yasuyuki Ohkawa. 2019. “A Chromatin Integration Labelling Method Enables Epigenomic Profiling with Lower Input.” Nature Cell Biology. https://doi.org/10.1038/s41556-018-0248-3.
- Harada, Akihito†, Kazumitsu Maehara†, Yusuke Ono, Hiroyuki Taguchi, Kiyoshi Yoshioka, Yasuo Kitajima, Yan Xie, et al. 2018. “Histone H3.3 Sub-Variant H3mm7 Is Required for Normal Skeletal Muscle Regeneration.” Nature Communications. https://doi.org/10.1038/s41467-018-03845-1.
- Maehara, Kazumitsu, and Yasuyuki Ohkawa. 2016. “Exploration of Nucleosome Positioning Patterns in Transcription Factor Function.” Scientific Reports. https://doi.org/10.1038/srep19620.
- Maehara, Kazumitsu†, Akihito Harada†, Yuko Sato, Masaki Matsumoto, Keiichi I. Nakayama, Hiroshi Kimura, and Yasuyuki Ohkawa. 2015. “Tissue-Specific Expression of Histone H3 Variants Diversified after Species Separation.” Epigenetics and Chromatin. https://doi.org/10.1186/s13072-015-0027-3.
†: co-first authors








