14-3-3タンパク質によるリン酸化シグナル経路の熱力学的・構造生物学的基盤
 研究代表者
研究代表者
大澤匡範
慶應義塾大学
http://square.umin.ac.jp/keio-skb/
研究概要
がんは、体の一部の細胞に生じた遺伝子変異をきっかけとして、正常細胞とは異なる自律的な増殖、周囲への浸潤、他臓器への遠隔転移によって、他の正常組織を破壊し、その機能を抑制し、血流・栄養・酸素などを奪い、体が衰弱し死に至る病気である。我が国には100万人以上のがん患者がおり、年間約40万人が亡くなるとされている(国立がん研究セ、2017年統計予測)。がんは日本人の死因の第一位であり、治療法の確立が急務である。多くのがん細胞においては、遺伝子変異によるRasタンパク質の活性化を起源として、その下流にあるキナーゼを活性化し、そのキナーゼは連鎖的に別のキナーゼや転写因子をリン酸化する。このようなリン酸化シグナルの連鎖と増幅が、がん細胞の増殖能や浸潤能を高める。
Ser/Thrのリン酸化状態は、14-3-3ζの結合により持続し、逆に14-3-3ζの結合阻害により抑制されることが分かってきた。したがって、がんにおけるリン酸化シグナルの数理モデルを確立するためには、14-3-3ζが結合する各キナーゼや転写因子(14-3-3ζのclientタンパク質)との結合親和性や、その基盤となる複合体の立体構造を明らかにする必要がある。
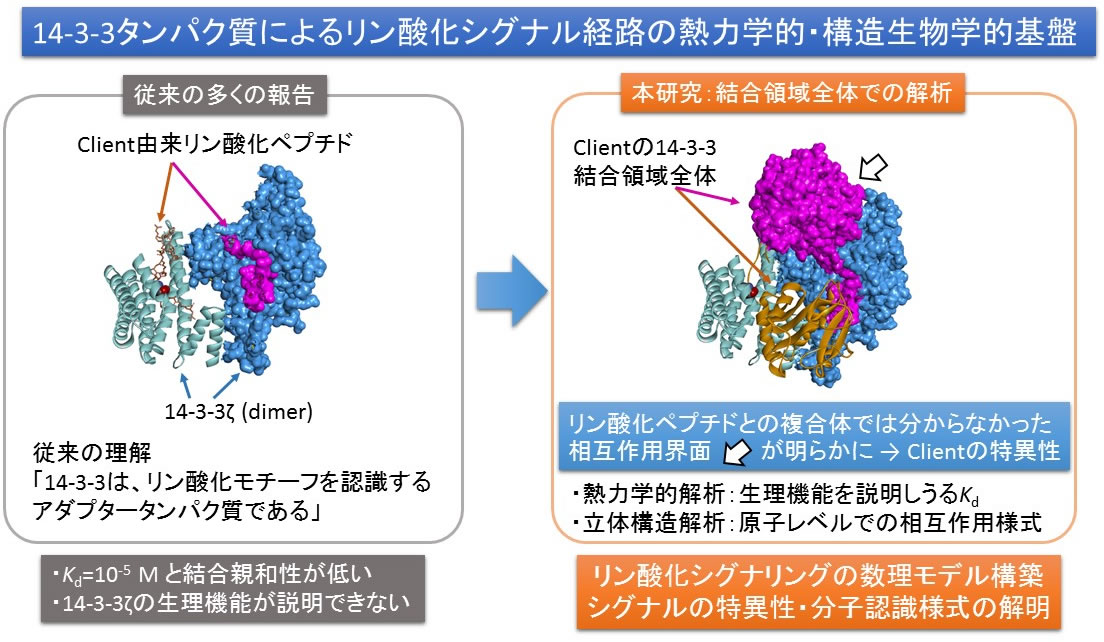
これまでに、14-3-3ζと、Ser/Thrがリン酸化されたペプチドとの結合親和性・複合体の立体構造が報告されている。しかし、その解離定数は10-5〜10-4 Mと大きく、その値では細胞生物学的な機能を説明できるほどの十分に高い親和性とは言えないことが問題であった。その原因として、従来の研究においては、リン酸化部位周辺のみを解析対象としたものがほとんどであり、それ以外の領域での相互作用の寄与が無視されていることが挙げられる。
そこで、本研究では、14-3-3ζのclientタンパク質全長、あるいは、14-3-3ζとの相互作用部位全体を用いた結合親和性の定量的解析、両者の複合体のX線結晶構造解析を行うことにより、リン酸化シグナルの数理モデルを構築する上での熱力学的・構造生物学的基盤を確立することを目的とする。さらに、この相互作用を調節するリガンドの作用機序を解明する。リン酸化部位以外の相互作用の寄与は、各client固有の性質が反映される可能性が高く、リン酸化部位の認識と単純化して捉えられてきた14-3-3ζの分子認識に対する考え方を一変させる重要な成果が期待できる。
参考文献
- Kano H., Toyama Y, Imai S, Iwahashi Y, Mase Y, Yokogawa M, Osawa M, and Shimada I. Structural mechanism underlying G protein family-specific regulation of G protein-gated inwardly rectifying potassium channel Nat Commun. (2019) in press
- Maruyama T, Imai S, Kusakizako T, Hattori M, Ishitani R, Nureki O, Ito K, Maturana AD, Shimada I, Osawa M. Functional roles of Mg2+ binding sites in ion-dependent gating of a Mg2+ channel, MgtE, revealed by solution NMR. eLIFE 7:e31596 (2018)
- Toyama Y, Kano H, Mase Y, Yokogawa M, Osawa M and Shimada I. Structural basis for the ethanol action on G-protein-activated inwardly rectifying potassium channel 1 revealed by NMR spectroscopy. Proc Natl Acad Sci USA 115 (15) 3858-3863 (2018).
- Sawazaki R, Imai S, Yokogawa M, Hosoda N, Hoshino S, Mio M, Mio K, Shimada I, Osawa M. Characterization of the multimeric structure of poly(A)-binding protein on a poly(A) tail. Sci. Rep. 8, 1455 (2018).
- Toyama Y, Mase Y, Kano H, Yokogawa M, Osawa M, Shimada I. "Nuclear magnetic resonance approaches for characterizing protein-protein interactions. Methods Mol Biol.1684, 115 (2018).
- Tomita A, Zhang M, Jin F, Zhuang W, Takeda H, Maruyama T, Osawa M, Hashimoto KI, Kawasaki H, Ito K, Dohmae N, Ishitani R, Shimada I, Yan Z, Hattori M, Nureki O. ATP-dependent modulation of MgtE in Mg2+ homeostasis. Nat Commun 8(1), 148 (2017).
- Toyama Y, Kano H, Mase Y, Yokogawa M, Osawa M, Shimada I. Dynamic regulation of GDP binding to G proteins revealed by magnetic field-dependent NMR relaxation analyses. Nat Commun 8, 14523 (2017).
- Nozaki T, Ozawa SI, Harada H, Kimura T, Osawa M, Shimada I. Disulfide mapping the voltage-sensing mechanism of a voltage-dependent potassium channel. Sci Rep 6, 37303 (2016).
- Toyama Y, Osawa M, Yokogawa M, and Shimada I. NMR Method for Characterizing Microsecond-to-Millisecond Chemical Exchanges Utilizing Differential Multiple-Quantum Relaxation in High Molecular Weight Proteins. J Am Chem Soc. 138(7), 2302-11(2016).
- Ozawa S, Kimura T, Nozaki T, Harada H, Shimada I, Osawa M. Structural basis for the inhibition of voltage-dependent K+ channel by gating modifier toxin. Sci Rep pp.14226(2015).








