オートファジーが関与する抗がん剤耐性獲得過程の数理モデル解析
 研究代表者
研究代表者
間木重行
東邦大学 医学部 生理学講座細胞生理学分野
https://www.toho-u.ac.jp/med/lab/lab_cytophysiology.html
研究概要
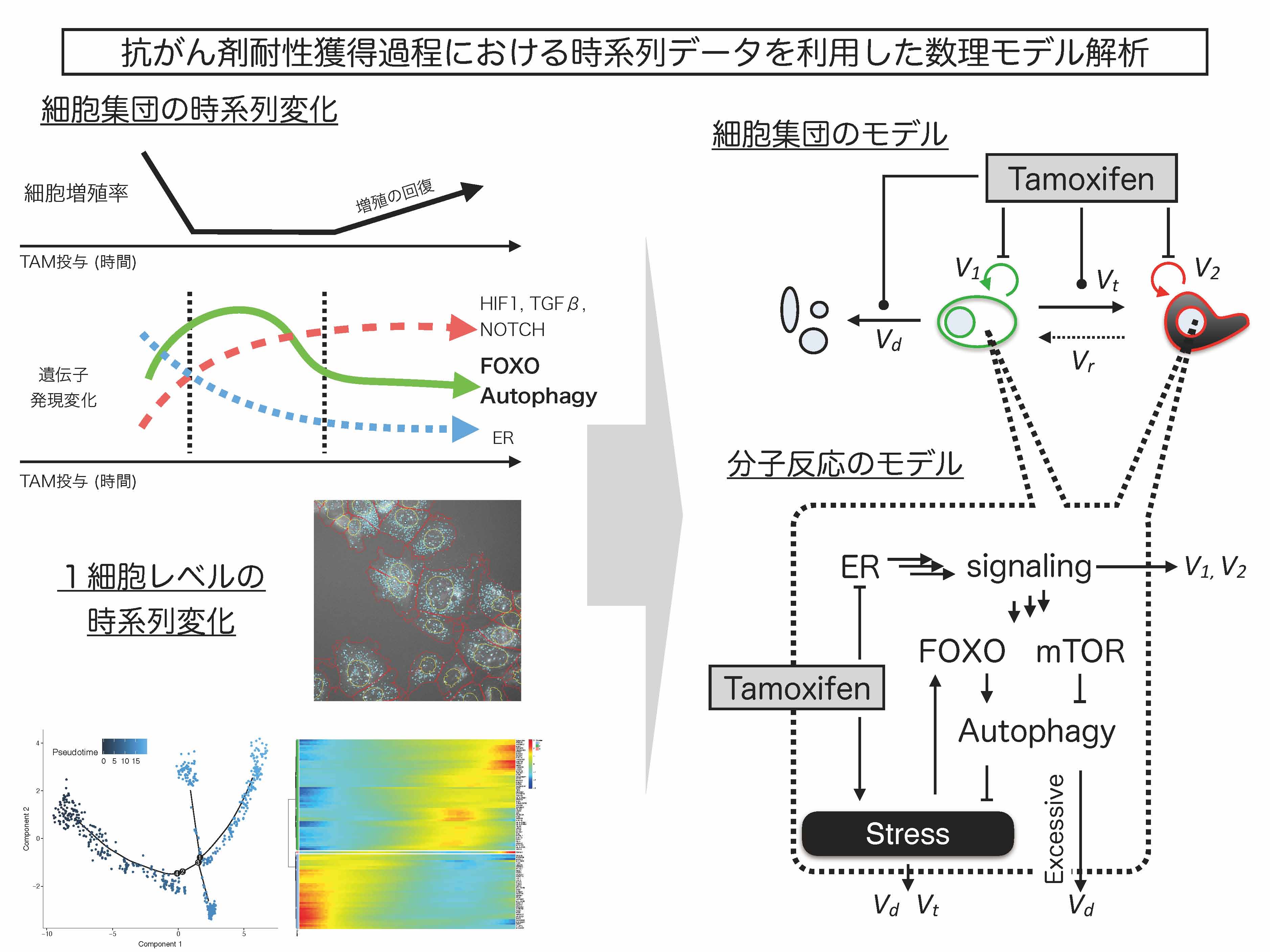
抗がん剤耐性はがん治療を困難にする原因の一つである。感受性株と耐性株の比較実験によって、エストロゲン受容体(ER)やmiRNAの発現変化、ERBB受容体シグナルやNFκBシグナル等代替シグナルの活性化など様々な現象が「耐性獲得の結果」起こっていることが明らかにされてきた。しかし、耐性が獲得される「途中過程」、即ち薬剤投与初期段階での細胞淘汰から耐性獲得期の増殖能力再獲得の一連の過程で働く詳細な分子機構は不明である。これまで私達は、ER陽性ヒト乳がん細胞が抗エストロゲン薬タモキシフェン(TAM)に対する耐性を獲得する過程の時系列遺伝子発現解析を行い、細胞が耐性を獲得する直前のタイミングで転写因子FOXOおよびオートファジー制御関連分子群が活性化することを見出した。
TAMよって誘導されるオートファジーが細胞死誘導に働く(Burch, et al., Carcinogenesis, 1996; Kohli, et al., Cancer Res., 2013など)か、細胞保護的(Thomas, et al., Breast Cancer Res. Treat., 2011; Lee et al., Autophagy, 2018など)かに関しては複数の報告が存在する。これら一見相反するかに見える先行研究の結果から、オートファジーが持つ細胞死・細胞生存の両方に貢献する機能の二面性やフィードバックを含む複雑な制御機構の存在が示唆される。従って、実験的アプローチのみによるTAM耐性獲得機構の全容解明は大きな困難を伴うことが予想される。そこで本研究では、オートファジーを始めとする耐性獲得の途中過程で働く分子制御に焦点をあて、それらが関与する細胞死/耐性獲得の運命選択の数理モデル構築を行う。構築されたモデルにおいてパラメータ感度等の解析を行うことで、TAM耐性獲得に著しく寄与する反応経路を明らかにし、実験的に検証する。将来的には、作用点の異なる抗がん剤耐性獲得機構にも解析対象を広げ、メカニズムの共通点・相違点を探っていきたい。
参考文献
- Magi S.*, Iwamoto K.*, Yumoto N, Hiroshima M., Nagashima T., Ohki R., Garcia-Munoz A., Volinsky N., Kriegsheim A.V., Sako Y., Takahashi K, Kimura S, Kholodenko B.N., Okada-Hatakeyama M. Transcriptionally inducible Pleckstrin homology-like domain family A member 1 attenuates ErbB receptor activity by inhibiting receptor oligomerization. J Biol. Chem., 293(6), 2206-2218, (2018)
- Magi S. *, Iwamoto K.*, Okada-Hatakeyama M., Current status of mathematical modeling of cancer – From the viewpoint of cancer hallmarks. Curr. Opin. Syst. Biol., 2, 38-47, (2017)
- Aitken S., Magi S., Alhendi. A. M. N., Itoh M., Kawaji H., Lassmann T., Daub CO., Arner E., Carninci P., Forrest A. R. R., Hayashizaki Y., FANTOM Consortium, Khachigian L.M., Okada-Hatakeyama M., Semple C. A.. Transcriptional Dynamics Reveal Critical Roles for Non-coding RNAs in the Immediate-Early Response. PLoS Comput. Biol. (4):e1004217 (2015)
- Carbajo D., Magi S., Itoh M., Kawaji H., Lassmann T., Arner E., Forrest A. R. R., Carninci P., Hayashizaki Y., Daub C.O., FANTOM Consortium, Okada-Hatakeyama M., Mar J. C. Application of Gene Expression Trajectories Initiated from ErbB Receptor Activation Highlights the Dynamics of Divergent Promoter Usage. PLoS ONE. 10(12):e0144176 (2015)
- Mina M.*, Magi S.*, Jurman G., Itoh M., Kawaji H., Lassmann T., Arner E., Forrest A. R. R., Carninci P., Hayashizaki Y., Daub C.O., FANTOM Consortium, Okada-Hatakeyama M., Furlanello C. Promoter-level expression clustering identifies time development of transcriptional regulatory cascades initiated by ErbB receptors in breast cancer cells. Sci. Rep. 5:11999 (2015)
- Magi S., Takemoto Y., Kobayashi H., Kasamatsu M., Akita T., Tanaka A., Takano K., Tashiro E., Igarashi Y. & Imoto M. 5-Lipoxygenase and cysteinyl leukotriene receptor 1 regulate epidermal growth factor-induced cell migration through Tiam1 upregulation and Rac1 activation. Cancer Sci. 105(3):290–6 (2014)
- Magi S., Saeki Y., Kasamatsu M., Tashiro E., Imoto M. Chemical genomic-based pathway analyses for epidermal growth factor-mediated signaling in migrating cancer cells. PLoS ONE. 9(5):e96776 (2014)
- Magi S.*, Shitara T*, Takemoto Y, Sawada M, Kitagawa M, Tashiro E, Takahashi Y., Imoto M. Novel derivatives of aclacinomycin A block cancer cell migration through inhibition of farnesyl transferase. J. Antibiot., 66, 165-170 (2013)
- Magi S., Tashiro E., Imoto M. A chemical genomic study identifying diversity in cell migration signaling in cancer cells. Sci Rep. 2:823 (2012)








