ヘリオロドプシンは何を伝えているのか?
 研究代表者
研究代表者
神取秀樹
名古屋工業大学大学院工学研究科
http://www.ach.nitech.ac.jp/~physchem/kandori/index_j.html
研究概要
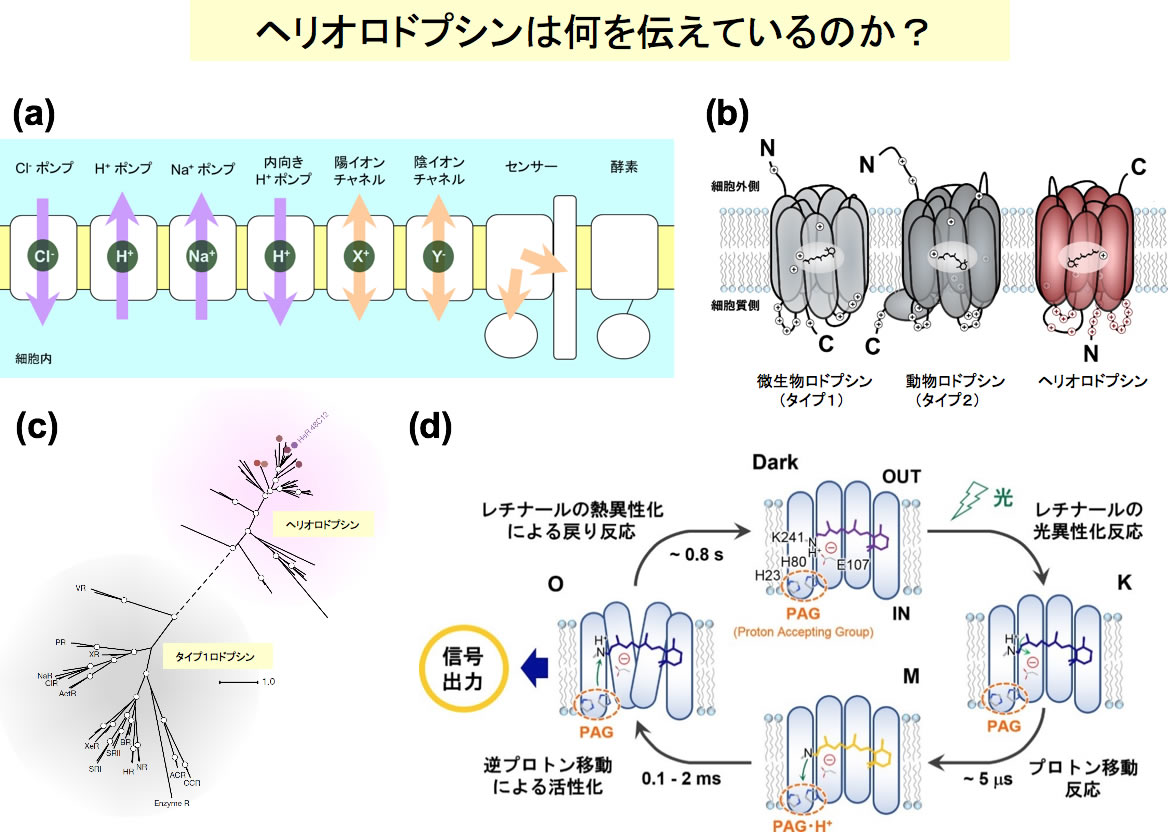
ロドプシンはレチナール分子を光吸収に用いる膜タンパク質である。動物が持つロドプシン(animal rhodopsin)は視物質としてGタンパク質を活性化する一方、微生物に含まれるロドプシン(microbial rhodopsin)はポンプ・チャネル・センサー・酵素など多彩な機能を持つ(図a)[1]。我々はこれまでロドプシンの構造機能相関を研究する中で、新しいロドプシン機能の発見や創成にも関わってきた[2-9]。ロドプシンは構造機能相関の研究が進んだ膜タンパク質としてよく知られているが、最近では光遺伝学(optogenetics)の主要ツールとして応用研究への期待も高い。
新しいロドプシンの機能が発見されてもそれらは配列相同性からすべて既知のロドプシンに分類され、地球上には微生物ロドプシン(タイプ1ロドプシン)と動物ロドプシン(タイプ2ロドプシン)しか存在しないと考えられていた。ところが2018年、我々はイスラエルとの国際共同研究により既知のロドプシンと相同性を持たないロドプシンを見出し、第三のロドプシンであるこのタンパク質群にヘリオロドプシンと名付けた(図b)[10]。一旦、ヘリオロドプシンが見つかると、500種類を超える相同タンパク質が真正細菌、古細菌、藻類などの真核生物やウイルスから見出され、ヘリオロドプシンは新しい世界を持つことがわかった(図c)。ヘリオロドプシンは膜トポロジーが反転しており(図b)、イオン輸送性を持たない。また相同性がほとんどないにも関わらず、既知の微生物ロドプシンとよく似た光反応の性質を示す(図d)。その機能に関して我々は光センサー機能を想定しているが、微生物由来のセンサーロドプシンと異なり、ヘリオロドプシンにはオペロンとなるような伝達タンパク質が存在しない。ヘリオロドプシンは細胞内に何を伝えているのであろうか? 本研究では、様々な実験手法を用いてヘリオロドプシンの機能解明を試みる。
参考文献
- Ernst, O. P., Lodowski, D. T., Elstner, M., Hegemann, P., Brown, L. S. and Kandori, H. "Microbial and animal rhodopsins: Structures, functions, and molecular mechanisms" Chem. Rev. 114, 126-163 (2014).
- Inoue, K., Ono, H., Abe-Yoshizumi, R., Yoshizawa, S., Ito, H., Kogure, K. and Kandori, H. "A light-driven sodium ion pump in marine bacteria" Nat. Commun. 4, 1678 (2013).
- Kato, H. E., Inoue, K., Abe-Yoshizumi, R., Kato, Y., Ono, H., Konno, M., Hososhima, S., Ishizuka, T., Hoque, M. R., Kunitomo, H., Ito, J., Yoshizawa, S., Yamashita, K., Takemoto, M., Nishizawa, T., Taniguchi, R., Kogure, K., Maturana, A. D., Iino, Y., Yawo, H., Ishitani, R., Kandori, H. and Nureki, O. "Structural basis for Na+ transport mechanism by a light-driven Na+ pump" Nature 521, 48-53 (2015).
- Inoue, K., Ito, S., Kato, Y., Nomura, Y., Shibata, M., Uchihashi, T., Tsunoda, S. P. and Kandori, H. "Natural light-driven inward proton pump" Nat. Commun. 7, 13415 (2016).
- Yoshida, K., Tsunoda, S. P., Brown, L. S. and Kandori, H. "A unique choanoflagellate enzyme rhodopsin exhibits light-dependent cyclic nucleotide phosphodiesterase" J. Biol. Chem. 292, 75314-7541 (2017).
- Kim, Y. S., Kato, H. E., Yamashita, K., Ito, S., Inoue, K., Ramakrishnan, C., Fenno, L. E., Evans, K. E., Paggi, J. M., Dror, R. O., Kandori, H., Kobilka, B. K. and Deisseroth, K. "Crystal structure of a natural anion-conducting channelrhodopsin GtACR1" Nature 561, 343-348 (2018).
- Kato, H. E., Kim, Y. S., Paggi, J. M., Evans, K. E., Allen, W. E., Richardson, C., Inoue, K., Ito, S., Ramakrishnan, C., Fenno, L. E., Yamashita, K., Hilger, D., Lee, S. Y., Berndt, A., Shen, K., Kandori, H., Dror, R. O., Kobilka, B. K. and Deisseroth, K. "Structural mechanisms of selectivity and gating in anion channelrhodopsins" Nature 561, 349-354 (2018).
- Bulzu, P. A., Andrei, A. Ş., Salcher, M. M., Mehrshad, M., Inoue, K., Kandori, H., Béjà, O., Ghai, R. and Banciu, H. L. "Casting light on Asgardarchaeota metabolism in a sunlit microoxic niche” Nat. Microbiol. in press (2019).
- Inoue, K., Marín, M. D. C., Tomida, S., Nakamura, R., Nakajima, Y., Olivucci, M. and Kandori, H. “Red-shifting mutation of light-driven sodium pump rhodopsin” Nat. Commun. in press (2019).
- Pushkarev, A., Inoue, K., Larom, S., Flores-Uribe, J., Singh, M., Konno, M., Tomida, S., Ito, S., Nakamura, R., Tsunoda, S. P., Philosof, A., Sharon, I., Yutin, N., Koonin, E. V., Kandori, H. and Béjà, O. “A distinct abundant group of microbial rhodopsins discovered using functional metagenomics” Nature 558, 595-599 (2018).








