多階層に跨る生体シグナル伝達システムの数理解析
 研究代表者
研究代表者
久保田浩行
九州大学 生体防御医学研究所
トランスオミクス医学研究センター
統合オミクス分野
http://www.bioreg.kyushu-u.ac.jp/labo/omics/
研究概要
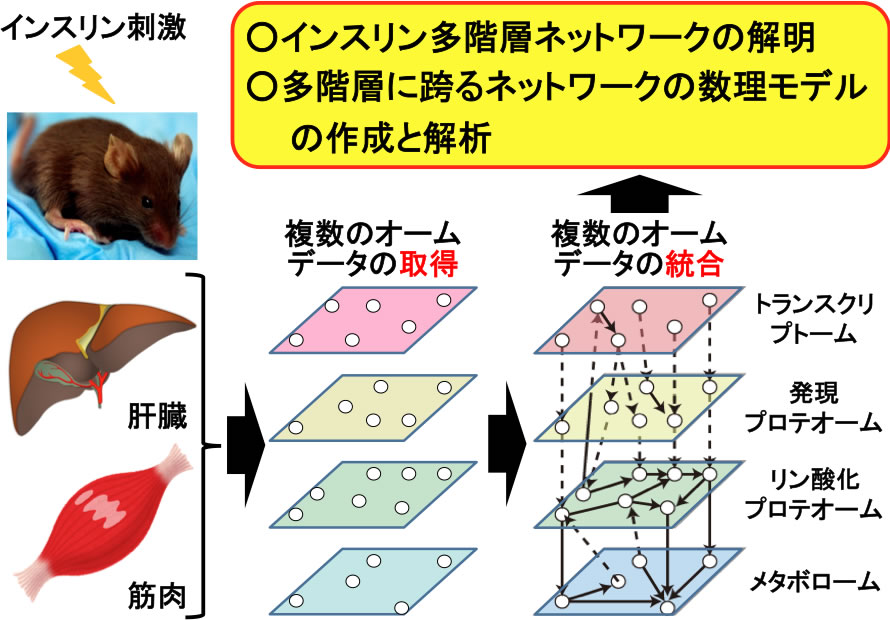
疾病を含む全ての生命現象は細胞内の遺伝子発現変化やタンパク質量、分子の複合体形成、翻訳後修飾、代謝変動など複数の「階層」に跨る数万の分子からなるシグナル伝達経路によって制御されている。つまり、多階層のネットワーク、そして、このネットワークによって伝達される動的なシグナルの特性(動的特性)を明らかにすることは疾病や生命現象の理解に直結する。このような解析を行うには従来の複数個の分子に注目してきた生物学的手法では手に負えず、細胞内の数万の分子の網羅的測定データから多階層ネットワークを再構築し、動的特性の解析を行うことのできる数理解析が必須となる。しかし、現在までに複数の網羅的データ(オームデータ)から多階層ネットワークを再構築し、数理モデルを作成することで生命現象の動的特性を明らかにした研究はない。
既に我々はAKTシグナル伝達経路に注目し、数理モデルを作成することでAKTシグナル伝達経路の動的特性を明らかにしてきた(Syst. Biol., 2013, Mol., Cell, 2012, Sci. Signal., 2010)。これらの研究により我々は、AKTシグナル伝達経路はインスリン刺激などの時間パターン依存的に下流の分子を選択的に制御できることを見出してきた。一方で我々は、世界に先駆けてリン酸化プロテオームとメタボロームのデータから多階層に跨るネットワークを再構築、新規経路を発見し、その動的特性も明らかにした(Cell Rep, 2015)。最近、我々を含めた多くの研究者がこのようなアプローチをトランスオミクス解析と呼び、今後増え続けるであろう網羅的なデータの新たな解析手法として注目している(Trends Biotech., 2016)。
そこで我々のグループでは、生体内におけるインスリン作用に注目する。初めに、個体(マウス)を用いて肝臓と筋肉におけるインスリン作用のマルチオームデータ(遺伝子発現、タンパク質発現、リン酸化タンパク質、代謝物)を取得、インスリン作用の多階層ネットワークを再構築する。インスリンシグナル経路は良く研究され理解されている多階層に跨る経路の一つであるが、その多階層に跨る全体像を同一実験下で取得・解析した研究は存在しない。次に、多階層に跨る実験データから数理モデルを作成することでその特性を理解する。このようなアプローチは生体内におけるインスリン作用の理解のみならず糖尿病などの疾患の理解に繋がることが期待される。
参考文献
- K. Yugi, H. Kubota, A. Hatano and S. Kuroda, “Trans-Omics: How To Reconstruct Biochemical Networks Across Multiple ‘Omic’ Layers” Trends Biotech., 34(4), 276-290 (2016)
- Y. Katsura, H. Kubota, K. Kunida, A. Kanno, S. Kuroda, T. Ozawa, “An optogenetic system for interrogating the temporal dynamics of Akt”. Sci. Rep., 5, 14589 (2015).
- K. Ohashi, H. Komada, S. Uda, H. Kubota, T. Iwaki, H. Fukuzawa, Y. Komori, M. Fujii, Y. Toyoshima, K. Sakaguchi, W. Ogawa, and S. Kuroda, “Glucose Homeostatic Law: Insulin Clearance Predicts the "Progression of Glucose Intolerance in Humans”. PLoS ONE, 10, e0143880 ( 2015).
- K. Yugi*, H. Kubota*, Y. Toyoshima, R. Noguchi, K. Kawata, Y. Komori, S. Uda, K. Kunida, Y. Tomizawa, Y. Funato, H. Miki, M. Matsumoto, K. I. Nakayama, K. Kashikura, K. Endo, K. Ikeda, T. Soga, and S. Kuroda, *Co-first author, “Reconstruction of insulin signal flow from phosphoproteome and metabolome data”. Cell Rep., 8, 1171-1183 (2014).
- Y. Akimoto, K. Yugi, S. Uda, Y. Komori, H. Kubota and S. Kuroda, “The extraction of simple relationships in growth factor-specific multiple-input and multipleoutput systems in cell-fate decisions by backward elimination PLS regression”. PLoS ONE, 8, e72780 (2013).
- S. Uda, T. Saito, T Kudo, T. Kokaji, T. Tsuchiya, H. Kubota, Y. Komori, Y. Ozaki, and S. Kuroda, “Robustness and compensation of information transmission of signaling pathways”. Science, 341, 558-61 (2013).
- R. Noguchi, H. Kubota, K. Yugi, Y. Toyoshima, Y. Komori, T. Soga, and S. Kuroda, “The Selective Control of Glycolysis, Gluconeogenesis and Glycogenesis by Temporal Insulin Patterns”. Mol. Syst. Biol.,9, 664 (2013).
- H. Kubota, R. Noguchi, Y. Toyoshima, Y. Ozaki, S. Uda, K. Watanabe, W. Ogawa and S. Kuroda, “Temporal Coding of Insulin Action through Multiplexing of the AKT Pathway”. Mol. Cell, 46, 820 (2012).
- K. Fujita, Y. Toyoshima, S. Uda, Y. Ozaki, H. Kubota, and S. Kuroda, “Decoupling of Receptor and Downstream Signals in the Akt Pathway by Its Low-Pass Filter Characteristics”. Science Signaling, 3, ra56 (2010).
- Y. Ozaki, S. Uda, T. Saito, J. Chung, H. Kubota, and S. Kuroda, “A quantitative image cytometry technique for time series or population analyses of signaling networks”. PLoS ONE, 5, e9955 (2010).








