数理解析に基づくMAPKシグナルと生命機能制御機構の解明
 研究分担者
研究分担者
石谷隆一郎
東京大学大学院理学系研究科・生物科学専攻
http://www.nurekilab.net/
研究概要
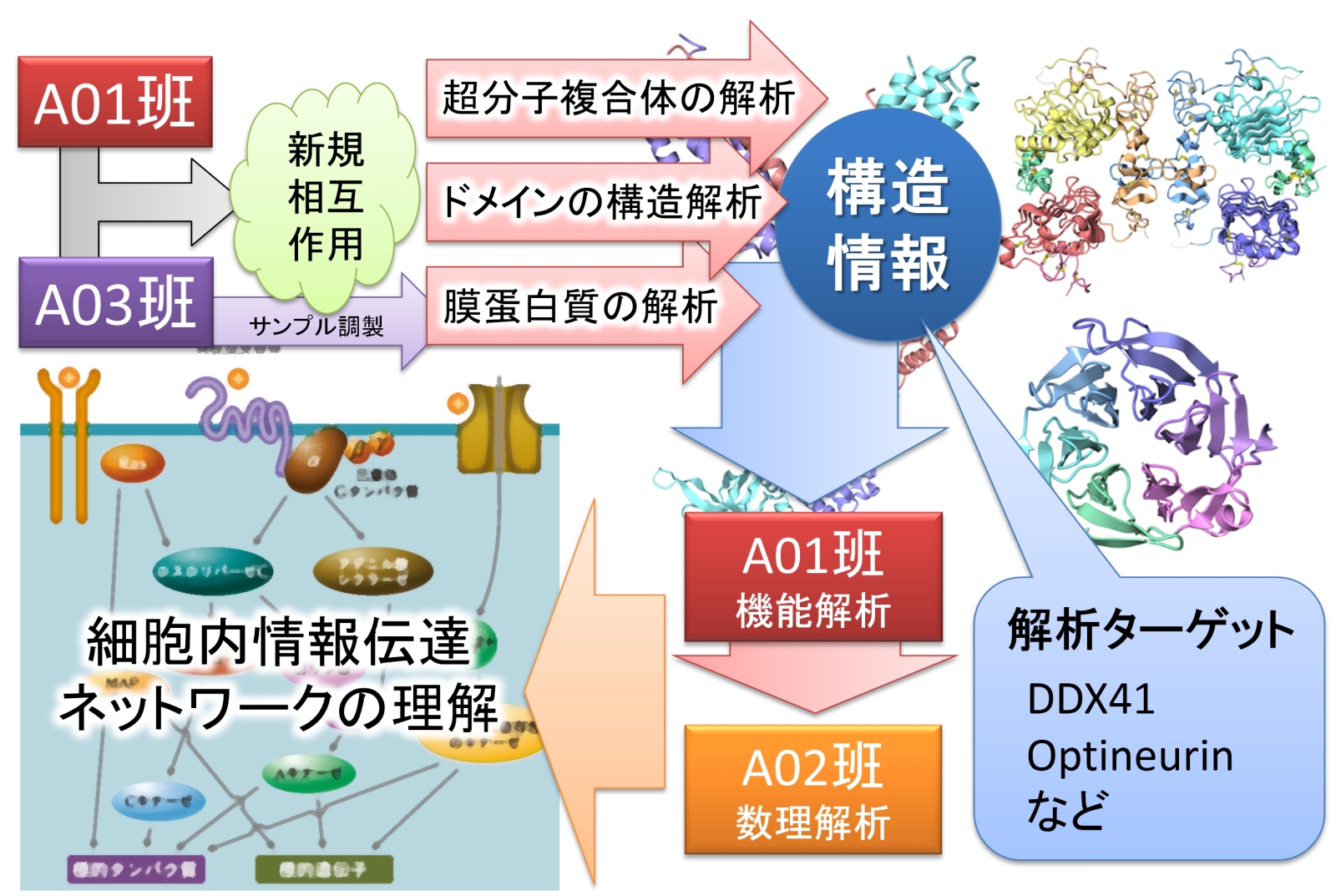
細胞内シグナル伝達は、蛋白質を含むさまざまな因子間の分子間相互作用により制御されており、癌、心血管疾患、神経変性疾患、自己免疫疾患から感染症に至るまでの、多くの疾病にも関与している。このシグナル伝達の全貌を解明するには、相互作用をネットワークとして捉え理解することが重要である。本計画では、このネットワークの理解に、構造生物学の手法を用いて3つの手法「1.各因子のドメインレベルの立体構造」、「2.高次複合体形成による相互作用の構造情報」、「3.細胞内ネットワークの入口としての膜受容体の構造情報」からアプローチする。
本計画では、まず「1.各因子のドメインレベルの立体構造」についてX線結晶構造解析の手法によりアプローチする。次に、「2.高次複合体形成による相互作用の構造情報」についてクライオ電子顕微鏡による単粒子解析法によりアプローチする。さらに、「3.細胞内ネットワークの入口としての膜受容体の構造情報」について、従来非常に困難であった膜蛋白質の結晶構造を多数解明してきた当研究室のノウハウを生かし、Gタンパク質共役型受容体等の膜受容体の構造解析を目指す。
申請者はこれまで、個々のシグナル伝達因子の構造解析を異分野との連携で行ってきたが、主に上記「1.ドメインレベルの相互作用」に着目したものであった。本計画では従来の研究をさらに発展させ、このマルチスケールの相互作用ネットワークを上記1〜3の手法から解明する。特にA01班・A03班が協働し新たに解明した新規相互作用を基軸に構造解析を行い、解明した構造情報をA01班研究者による細胞生物学的実験や、A02班研究者による数理モデルシミュレーションへとフィードバックし、構造・機能の両面から細胞情報ネットワークの解明にアプローチする。本計画では、まずは既に連携が開始しているターゲット、RANK-TRAF6経路に関わる因子(井上班)、MAPK経路に関わる因子(武川班)やLUBACやDDX41(井上・徳永班)の構造解析を、計画班間との協働で遂行するが、他計画班や公募班で新規に同定された因子に関しても同様の戦略にて構造解析を行う。また、当研究室で進行中のプロジェクトも推進する。以上より、領域内研究者と緊密な連携をはかり領域全体の推進に貢献する。
参考文献
- Crystal structure of the plant receptor-like kinase TDR in complex with the TDIF peptide.
Morita J, Kato K, Nakane T, Kondo Y, Fukuda H, Nishimasu H, Ishitani R, Nureki O.
Nat Commun. 2016 Aug 8;7:12383. doi: 10.1038/ncomms12383. - Structural and functional insights into IZUMO1 recognition by JUNO in mammalian fertilization.
Kato K, Satouh Y, Nishimasu H, Kurabayashi A, Morita J, Fujihara Y, Oji A, Ishitani R, Ikawa M, Nureki O.
Nat Commun. 2016 Jul 15;7:12198. doi: 10.1038/ncomms12198. - Structural basis for amino acid export by DMT superfamily transporter YddG.
Tsuchiya H, Doki S, Takemoto M, Ikuta T, Higuchi T, Fukui K, Usuda Y, Tabuchi E, Nagatoishi S, Tsumoto K, Nishizawa T, Ito K, Dohmae N, Ishitani R, Nureki O.
Nature. 2016 May 30;534(7607):417-20. doi: 10.1038/nature17991. - Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA.
Yamano T, Nishimasu H, Zetsche B, Hirano H, Slaymaker IM, Li Y, Fedorova I, Nakane T, Makarova KS, Koonin EV, Ishitani R, Zhang F, Nureki O.
Cell. 2016 May 5;165(4):949-62. doi: 10.1016/j.cell.2016.04.003. Epub 2016 Apr 21. - Structural basis for specific inhibition of Autotaxin by a DNA aptamer.
Kato K, Ikeda H, Miyakawa S, Futakawa S, Nonaka Y, Fujiwara M, Okudaira S, Kano K, Aoki J, Morita J, Ishitani R, Nishimasu H, Nakamura Y, Nureki O.
Nat Struct Mol Biol. 2016 May;23(5):395-401. doi: 10.1038/nsmb.3200. Epub 2016 Apr 4. - Structural Basis for the Altered PAM Specificities of Engineered CRISPR-Cas9.
Hirano S, Nishimasu H, Ishitani R, Nureki O.
Mol Cell. 2016 Mar 17;61(6):886-94. doi: 10.1016/j.molcel.2016.02.018. - Structure and biological function of ENPP6, a choline-specific glycerophosphodiester-phosphodiesterase.
Morita J, Kano K, Kato K, Takita H, Sakagami H, Yamamoto Y, Mihara E, Ueda H, Sato T, Tokuyama H, Arai H, Asou H, Takagi J, Ishitani R, Nishimasu H, Nureki O, Aoki J.
Sci Rep. 2016 Feb 18;6:20995. doi: 10.1038/srep20995. - Structure and Engineering of Francisella novicida Cas9.
Hirano H, Gootenberg JS, Horii T, Abudayyeh OO, Kimura M, Hsu PD, Nakane T, Ishitani R, Hatada I, Zhang F, Nishimasu H, Nureki O.
Cell. 2016 Feb 25;164(5):950-61. doi: 10.1016/j.cell.2016.01.039. Epub 2016 Feb 11. - Outward- and inward-facing structures of a putative bacterial transition-metal transporter with homology to ferroportin.
Taniguchi R, Kato HE, Font J, Deshpande CN, Wada M, Ito K, Ishitani R, Jormakka M, Nureki O.
Nat Commun. 2015 Oct 13;6:8545. doi: 10.1038/ncomms9545. - Crystal Structure of Staphylococcus aureus Cas9.
Nishimasu H, Cong L, Yan WX, Ran FA, Zetsche B, Li Y, Kurabayashi A, Ishitani R, Zhang F, Nureki O.
Cell. 2015 Aug 27;162(5):1113-26. doi: 10.1016/j.cell.2015.08.007.








