高精度プロテオミクスによるシグナル伝達制御機構の数理システム解析
 研究代表者
研究代表者
尾山大明
東京大学医科学研究所 疾患プロテオミクスラボラトリー
http://www.ims.u-tokyo.ac.jp/mpl/top.html
 研究分担者
研究分担者
秦 裕子
東京大学医科学研究所 疾患プロテオミクスラボラトリー
研究概要
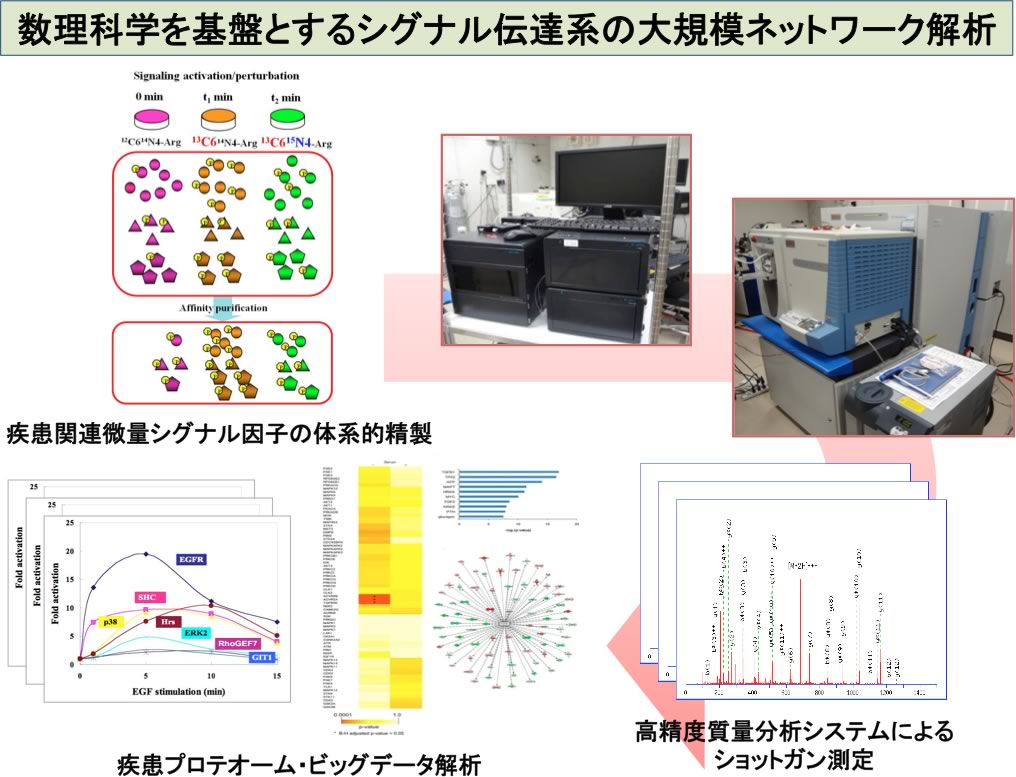
シグナル伝達系は細胞の運命決定を担う最も重要な生命制御システムの一つであり、リン酸化やユビキチン化・アセチル化をはじめとするタンパク質翻訳後修飾による精細な制御に基づく複雑な相互作用ネットワークによって、転写・翻訳をはじめとする基本的な生命活動の駆動力として機能している。がんをはじめとする多くの疾患はこの複雑な生命システムの制御異常・破綻を起点としていることから、各種疾患に関与する情報伝達ネットワークの異常を理論的に制御するためには、シグナル伝達系を構成しているタンパク質群やそれらが織り成す複合体に関する包括的な理解のみならず、翻訳後修飾による微細な制御を包含した統合的なネットワーク解析を通してシステムとしての作動原理を明らかにすることが必要不可欠である。
本研究課題では、細胞外部からの刺激入力や阻害剤による摂動によって変動するシグナル複合体やそれらのリン酸化、ユビキチン化、アセチル化等の翻訳後修飾レベルを網羅的・統合的に計測する高深度定量プロテオーム解析基盤を確立すると共に、得られる大規模な同定・定量情報に基づいて鍵となるシグナル制御を統計科学的に抽出する情報解析方法論を構築し、分子細胞生物学的手法による詳細な機能解析、並びに数理モデルによる精密な反応制御パラメータ解析と相互に連携するオミクス情報解析基盤の構築を行う。
現在までにMAPK/NF-κB経路をはじめとして多くの疾患関連シグナル因子が発見されているが、これらの因子が実際に細胞中で機能を担う際のアミノ酸修飾構造の変化や相互作用に関する動態変化を精密に捉えることによって、理論的な薬剤の設計・創出や根本的な治療への展開が可能になると考えられる。本研究計画で計測される大規模な質量分析データには大量の新規翻訳後修飾部位や相互作用因子に関する同定・定量データを含むことが想定されることから、これらの「プロテオーム・ビッグデータ」に基づくシグナルネットワーク解析手法の開発を通して、シグナル伝達系の異常に起因する疾患を対象とした薬剤ターゲットの理論的な探索に関する新たな研究体系の創成が可能となる。
参考文献
- Narushima Y, Kozuka-Hata H, Tsumoto K, Inoue J, and Oyama M.
Quantitative phosphoproteomics-based molecular network description for high-resolution kinase-substrate interactome analysis. Bioinformatics, 32: 2083-2088 (2016). - Narushima Y, Kozuka-Hata H, Koyama-Nasu R, Tsumoto K, Inoue J, Akiyama T, and Oyama M.
Integrative Network Analysis Combined with Quantitative Phosphoproteomics Reveals Transforming Growth Factor-beta Receptor type-2 (TGFBR2) as a Novel Regulator of Glioblastoma Stem Cell Properties. Mol. Cell. Proteomics, 15: 1017-1031 (2016). - Hirano A, Yumimoto K, Tsunematsu R, Matsumoto M, Oyama M, Kozuka-Hata H, Nakagawa T, Lanjakornsiripan D, Nakayama KI, and Fukada Y.
FBXL21 regulates oscillation of the circadian clock through ubiquitination and stabilization of cryptochromes. Cell, 152: 1106-1118 (2013). - Kozuka-Hata H, Nasu-Nishimura Y, Koyama-Nasu R, Ao-Kondo H, Tsumoto K, Akiyama T, and Oyama M.
Phosphoproteome of human glioblastoma initiating cells reveals novel signaling regulators encoded by the transcriptome. PLoS One, 7: e43398 (2012). - Oyama M, Nagashima T, Suzuki T, Kozuka-Hata H, Yumoto N, Shiraishi Y, Ikeda K, Kuroki Y, Gotoh N, Ishida T, Inoue S, Kitano H, and Okada-Hatakeyama M.
Integrated quantitative analysis of the phosphoproteome and transcriptome in tamoxifen-resistant breast cancer. J. Biol. Chem., 286: 818-829 (2011). - Arii J, Goto H, Suenaga T, Oyama M, Kozuka-Hata H, Imai T, Minowa A, Akashi H, Arase H, Kawaoka Y, and Kawaguchi Y.
Non-muscle myosin IIA is a functional entry receptor for herpes simplex virus 1. Nature, 467: 859-862 (2010). - Tasaki S, Nagasaki M, Kozuka-Hata H, Semba K, Gotoh N, Hattori S, Inoue J, Yamamoto T, Miyano S, Sugano S, and Oyama M.
Phosphoproteomics-based modeling defines the regulatory mechanism underlying aberrant EGFR signaling. PLoS One, 5: e13926 (2010). - Oyama M, Kozuka-Hata H, Tasaki S, Semba K, Hattori S, Sugano S, Inoue J, and Yamamoto T.
Temporal perturbation of tyrosine-phosphoproteome dynamics reveals the system-wide regulatory networks. Mol. Cell. Proteomics, 8: 226-231 (2009). - Oyama M, Kozuka-Hata H, Suzuki Y, Semba K, Yamamoto T, and Sugano S.
Diversity of translation start sites may define increased complexity of the human short ORFeome. Mol. Cell. Proteomics, 6: 1000-1006 (2007). - Oyama M, Itagaki C, Hata H, Suzuki Y, Izumi T, Natsume T, Isobe T, and Sugano S.
Analysis of small human proteins reveals the translation of upstream open reading frames of mRNAs. Genome Res., 14: 2048-2052 (2004).








