自然免疫シグナルにおけるユビキチン化基質の網羅的同定と解析
 研究代表者
研究代表者
渡部 昌
北海道大学 大学院医学研究院 生化学分野
https://hokudai-ikagaku.jp/
研究概要
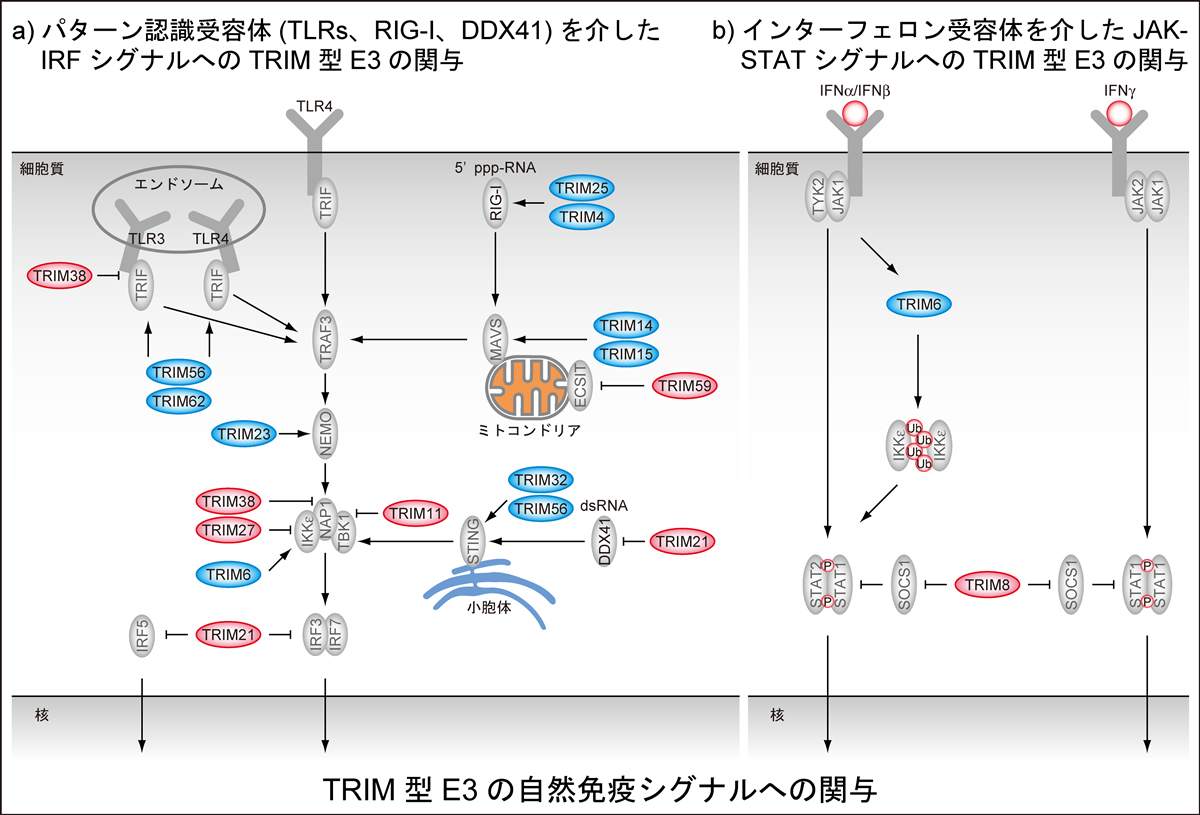
ユビキチンによるタンパク質の可逆的修飾は免疫・癌・感染をはじめとした様々な生命現象を支える極めて重要な翻訳後修飾の一つであり、ユビキチンリガーゼ(E3)が選択的に基質を認識することによって起こる反応である。したがって、個々のE3に特異的な基質を同定しそのユビキチン化部位を決定することは様々な生命現象を理解する上で重要となる。私達はこれまで、TRIM型E3の基質の同定およびその機能の解析を中心に研究を行ってきた。この中で、他のグループの報告も含め数多くのTRIM型E3が自然免疫シグナルの制御に重要な役割を果たしていることが明らかとなってきている(図)。
一方、多くの研究者のこれまでの取組みにも関わらず、膨大なE3酵素の数に比較してその基質同定の成功例は少なく、検証に多大な時間を要するのが現状であった。私達も、これまでにE3と基質の結合力を利用した方法やレポーターアッセイを指標としたスクリーニング法により基質の同定を試み、細胞における機能解析を行ってきた。しかしE3と結合力の強い分子はユビキチン化を受けないことが多いことや、偽陽性が多いことなどの特異度の低さが大きな問題であった。基質の半減期を指標として網羅的に同定する方法なども報告されているが、プロテアソーム輸送シグナル以外のユビキチン化を受ける基質は同定できないという問題点があった。近年、Toczyskiらによってユビキチン結合ドメイン(UBA)とE3の融合プローブによるLigase trap法(Mol Cell, 2014)が、また吉田らによって、ユビキチン高親和性ドメイン(TUBE)とユビキチン化を受けたペプチドを認識するK-εGG抗体を組み合わせるTR-TUBE法(PNAS, 2015)が提唱された。私達は、これらの手法の長所を組み合わせていくつかの改良を加えた基質同定法の開発を試みており、感度のさらなる向上に成功している。
E3は高い特異性で基質をユビキチン化するが、その基質は単一ではなく複数存在することが多い。しかし一方で、過去の報告では個々のE3と基質との関係が解析されているのみであり、E3がシグナル伝達の中で複数存在する基質のうち、どの基質にどの程度のユビキチンを付加しているのかに関する網羅的且つ定量的な情報は解析されていない。以上のことから、本研究では、TRIM型E3が特に重要な役割を果たすと考えられる①パターン認識受容体(TLRs、RIG-I、DDX41)を介したIRFシグナルと、②インターフェロン受容体を介したJAK-STATシグナルに着目し、これらのシグナル伝達に関与するTRIM型E3の基質を新規手法によって網羅的に同定し定量することで、数理解析へ展開するための基盤を構築したい。
参考文献
- Watanabe M and Hatakeyama S: TRIM proteins and diseases. J. Biochem., 161, 135-144, 2017.
- Mizushima W, Takahashi H, Watanabe M, Kinugawa S, Matsushima S, Takada S, Yokota T, Furihata T, Matsumoto J, Tsuda M, Chiba I, Nagashima S, Yanagi S, Matsumoto M, Nakayama K, Tsutsui H, Hatakeyama S: The novel heart-specific RING finger protein 207 is involved in energy metabolism in cardiomyocytes. J. Mol. Cell. Cardiol., 100, 43-53, 2016.
- Anwar D, Takahashi H, Watanabe M, Suzuki M, Fukuda S and Hatakeyama S: p53 represses the transcription of snRNA genes by preventing the formation of little elongation complex. Biochim. Biophys. Acta-Gene Regul. Mech., 1859, 975-982, 2016
- Suzuki M, Watanabe M, Nakamaru Y, Takagi D, Takahashi H, Fukuda S and Hatakeyama S: TRIM39 negatively regulates the NFκB-mediated signaling pathway through stabilization of cactin. Cell. Mol. Life Sci., 73, 1085-1101, 2016
- Watanabe M, Takahashi H, Saeki Y, Ozaki T, Itoh S, Suzuki M, Mizushima W, Tanaka K, Hatakeyama S: The E3 ubiquitin ligase TRIM23 regulates adipocyte differentiation via stabilization of the adipogenic activator PPARγ. eLife, 4, e05615, 2015.
- Takahashi H, Takigawa I, Watanabe M, Anwar D, Shibata M, Tomomori-Sato C, Sato S, Ranjan A, Seidel CW, Tsukiyama T, Mizushima W, Hayashi M, Ohkawa Y, Conaway JW, Conaway RC, Hatakeyama S: MED26 regulates the transcription of snRNA genes through the recruitment of little elongation complex. Nat. Commun., 6, 5941, 2015








