数理解析に基づくmRNA分解を介した概日リズム制御機構の解明
 研究代表者
研究代表者
高橋明格
沖縄科学技術大学院大学 細胞シグナルユニット
https://groups.oist.jp/ja/csu
研究概要
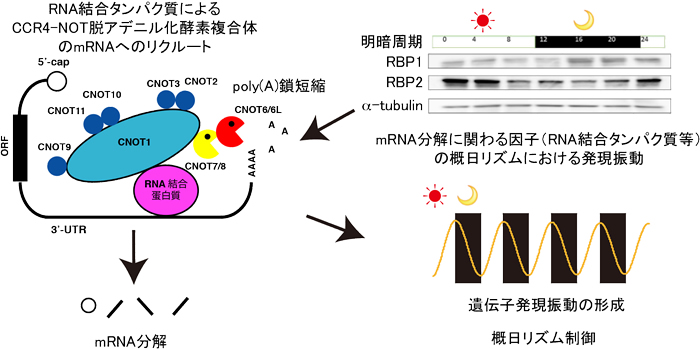
概日リズムは、生物に備わった約24時間周期のリズムであり、睡眠覚醒、ホルモン分泌、体温、心拍、高次脳機能を制御する。この生理現象は、リミットサイクルと呼ばれる、摂動に対してロバストに周期活動を継続するダイナミクスにより形成され、遺伝子発現自体も振動することから、数理的解析が多くなされてきた。mRNA量は、転写合成とmRNA分解により厳密に制御されている。これまでの遺伝子発現制御機構は転写を中心に解析が進められてきたが、概日リズムにおいては、遺伝子発現の振動形成の78%が転写制御では説明できず、mRNA分解を始めとする転写後調節の必要性が示唆されている。よって、概日リズムにおけるmRNA分解を介した遺伝子発現制御の数理モデルの確立、分子機構の解明は重要な課題である。
多くのmRNAは、3’末端のpoly(A)鎖が短縮し取り除かれた後、5’キャップ構造がデキャピング酵素により外され、5’および3’両端から特異的なexonucleaseにより分解される。このため、poly(A)鎖の短縮(脱アデニル化)は、mRNA分解の開始ステップと言える。CCR4-NOT複合体は、真核生物の主要な脱アデニル化酵素である。CCR4-NOT複合体は2 MDaの複合体であり、少なくとも11個のサブユニット(Cnot1-3, 6l, 6-11)で構成される。このうち、Cnot6/6l/7/8は脱アデニル化酵素活性を持ち、また、Cnot1は巨大な足場タンパクであり、すべてのサブユニットをつなぎとめる。このため、Cnot1はCCR4-NOT複合体の脱アデニル化活性の制御に重要な役割を担う。これまでに、我々は、各サブユニットの遺伝子欠損マウスを用いて、CCR4-NOT複合体がエネルギー代謝、精子形成などの多くの生命現象に関わることを示してきた(Nakamura et al., 2004, Nature Genet; Morita et al., 2011, EMBO J; Takahashi et al., 2015, Cell Rep)。さらに、興味深いことに、近年の研究で、Cnot1遺伝子ヘテロ欠損マウスが概日リズムの遅延を呈することを明らかにしてきた。また、CCR4-NOT複合体のサブユニットの一部やmRNA分解に重要なRNA結合タンパク質(RBP)が概日リズムにおいて、その発現を振動させるという知見も得ており、mRNA分解の概日リズム制御における重要性が示唆される。そこで、本研究では、視交叉上核および肝臓における、CCR4-NOT複合体ならびにRNA結合タンパク質によるmRNA分解を介した概日リズム制御の詳細な分子基盤を明らかにする。さらに、研究連携者、山口大学医学部浅井義之教授の協力のもと、PhysioDesignerならびにFlintを用いて、CCR4-NOT複合体・RNA結合タンパク質によるmRNA分解を中心とした遺伝子発現制御系の数理モデルを多階層的に構築し、概日リズムにおける遺伝子発現の振動をシミュレーションする。また、mRNA分解と転写・翻訳とのクロストークを評価することで、概日リズム制御におけるmRNA分解の重要性と妥当性を明らかにする。
参考文献
- Li, X., Morita, M., Kikuguchi, C., Takahashi, A., Suzuki, T., Yamamoto, T. (2017). Adipocyte-specific disruption of mouse Cnot3 causes lipodystrophy. FEBS Lett. 591, 358-368.
- Zukeran, A., Takahashi, A. *, Takaoka, S., Mohamed, HM., Suzuki, T., Ikematsu, S., Yamamoto, T. (2016). The CCR4-NOT deadenylase activity contributes to generation of induced pluripotent stem cells. Biochem. Biophys. Res. Commun. 474, 233-239. *; Corresponding author
- Takahashi, A., Adachi, S., Morita, M., Tokumasu, M., Natsume, T., Suzuki, T., Yamamoto, T. (2015). Post-Transcriptional Stabilization of Ucp1 mRNA Protects Mice from Diet-Induced Obesity. Cell Rep. 13, 2756-2767.
- Shirai, Y.T., Suzuki, T., Morita, M., Takahashi, A., Yamamoto, T. (2014). Multifunctional roles of the mammalian CCR4-NOT complex in physiological phenomena. Front. Genet. 5, 286 doi: 10.3389/fgene.2014.00286.
- Takahashi, A., Morita, M., Yokoyama, K., Suzuki, T., Yamamoto, T. (2012). Tob2 inhibits PPARg2 expression by sequestering Smads and C/EBPa during adipocyte differentiation. Mol. Cell. Biol. 32, 5067-5077.
- Takahashi, A., Kikuguchi, C., Morita, M., Shimodaira, T., Tokai-Nishizumi, N., Yokoyama, K., Ohsugi, M., Suzuki, T., Yamamoto, T. (2012). Involvement of CNOT3 in mitotic progression through inhibition of MAD1 expression. Biochem. Biophys. Res. Commun. 419, 268-273.
- Ito, K., Takahashi, A., Morita, M., Suzuki, T., Yamamoto, T. (2011). The role of the CNOT1 subunit of the CCR4-NOT complex in mRNA deadenylation and cell viability. Protein Cell 2, 755-763.
- Chen, C., Ito, K., Takahashi, A., Wang, G., Suzuki, T., Nakazawa, T., Yamamoto, T., Yokoyama, K. (2011). Distinct expression patterns of the subunits of the CCR4-NOT deadenylase complex during neural development. Biochem. Biophys. Res. Commun. 411, 360-364.
- Takahashi, A., Obata, Y., Fukumoto, Y., Nakayama, Y., Kasahara, K., Kuga, T., Higashiyama, Y., Saito, T., Yokoyama, K.K., Yamaguchi, N. (2009). Nuclear localization of Src-family tyrosine kinases is required for growth factor-induced euchromatinization. Exp. Cell Res. 315, 1117-1141.
- Yamasaki, T., Takahashi, A., Pan, J., Yamaguchi, N., Yokoyama, K.K. (2009). Phosphorylation of Activation Transcription Factor-2 at Serine 121 by Protein Kinase C Controls c-Jun-mediated Activation of Transcription. J. Biol. Chem. 284, 8567-8581.








