1細胞ダイナミクスと相空間モデルによる情報処理ネットワーク解析法の開発
 研究代表者
研究代表者
佐甲靖志
理化学研究所
http://www.riken.go.jp/cell-info/
研究概要
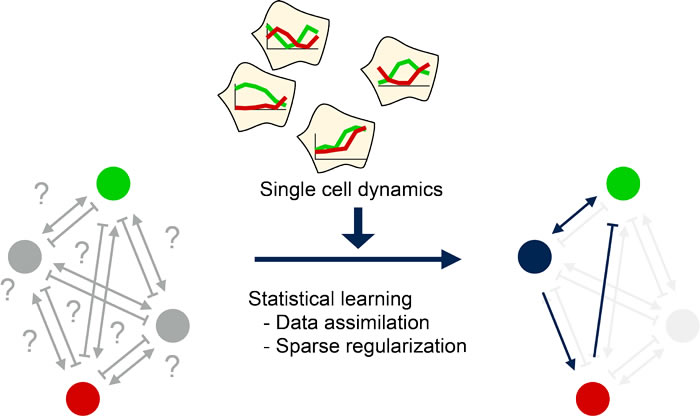
単一細胞の反応ダイナミクスに基づいて細胞内情報処理反応ネットワークを逆行分析し、その動態を数理的に再現・理解・予測するためのモデル構築法を開発する。従来の多くの反応ネットワークモデルは個別の分子反応の知識を積み上げて組み立てられているが、情報の不足による不確実性を免れない。本研究では、複数・並列の単一細胞反応動態の実測値に基づいて反応ネットワークを統計的に推定し、「実効的に最小・充分の数理モデル」を作る。
具体的には細胞運命決定システムであるErbB-RAS-MAPKネットワークを解析し、新しい方法の有効性を検証する。細胞内反応ネットワークを数理モデルとして記述して挙動を再現し、細胞の振舞いを理解・予測することは、数理生物学の重要な課題の一つであるが、従来研究の多くは、個別の分子反応に関する知識を積み上げて反応ネットワークを構築し、連立微分方程式として記述して、挙動を解いてきた。そのような研究の問題点として、反応システムに関する我々の知識不足のために、構成されたネットワークも不完全で恣意的なものにならざるを得ず、たとえば、研究の進捗状況に依存して、反応記述の精度がひとつのネットワーク内の部分によって異なること。分子の濃度・密度、反応速度定数といった、個々の反応に関するパラメータが(特に細胞内において)ほとんど実測されておらず、少数の分子反応ダイナミクスを(不完全なモデルに基づいて)再現するように推定されているにすぎないこと。その他、モデル構築やパラメータ推定の条件となる反応ダイナミクスの実測値が、多くの場合複数細胞の平均値であって、単一細胞の挙動とは異なる可能性があること。などが上げられる。
以上のような問題にもかかわらず従来手法は一定の成功を収めてきたが、一方で、従来手法を補完するため、単一細胞で実測した反応ダイナミクスから、余分な仮定をできるだけ排除して、データ依存的に実効反応ネットワークとそのパラメータを推定する方法が必要である。両方法の比較によって、細胞挙動の再現・理解・予測がより確実になることを期待する。
本研究では、細胞内反応の複数時系列を連立微分方程式に基づく相空間モデルで記述し、多数の1細胞時系列データに粒子フィルターを使って統計的に実験データを再現する。その際、要素間を繋ぐネットワーク自体も冗長なモデルから出発して、正規化によって余分な経路を削除しながら、ネットワークとパラメータの同時推定を行う。人工遺伝子発現回路などを題材として計算法開発と検証を行い。その後、Erb-RAS-MAPK回路の解析を進める。
参考文献
- Arata, Y., et al. (2016) Cortical polarity of the RING protein PAR-2 is maintained by exchange rate kinetics at the cortical-cytoplasm boundary. Cell Rep. 16, 2156-2168.
- Nakamura, Y., Hibino, K., Yanagida, T., and Sako, Y. (2016) Switching of the positive feedback for RAS activation by a concerted function of SOS membrane association domains. Biophys. Physicobiol. 13, 1-11.
- Shindo, Y., et al. (2016) Conversion of graded into switch-like nuclear translocation via autoregulatory mechanisms in ERK signaling. Nat. Comm. 7, 10485 (1-10).
- Shinohara, H., et al. (2014) Positive feedback within a kinase signaling complex functions as a switch mechanism for NF-kB activation. Science, 344, 760-764.
- Mouri, K. and Sako, Y. (2013) Optimality conditions for cell-fate heterogeneity that lead to the ability to maximize effects of growth factors in PC12 cells. PLoS Comp. Biol. 9, e1003320 (1-15).








