キナーゼ間クロストークの解明へ向けたキナーゼドメイン間相互作用の構造基盤の解明
 研究代表者
研究代表者
小橋川敬博
熊本大学 大学院生命科学研究部 (薬)
http://www.pharm.kumamoto-u.ac.jp/Labs/bunseki/member.html
研究概要
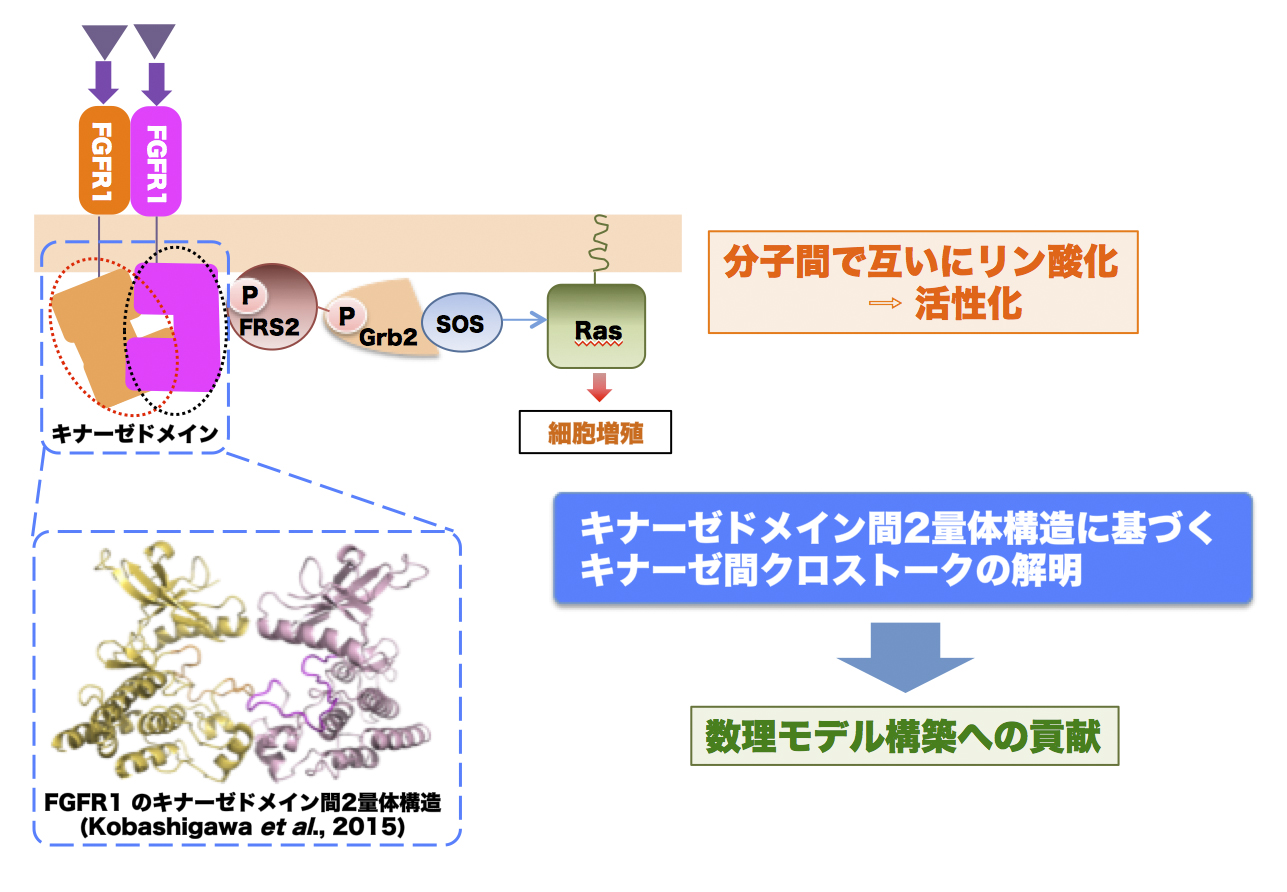
シグナル伝達の数理モデルを構築する上でシグナル伝達ネットワークの解明が重要です。未だ未解明な点が多いチロシンキナーゼ間のクロストークが解明できれば、数理モデル構築に大きく貢献すると考えられます。本研究課題ではキナーゼドメイン間の過渡的2量体の構造基盤に基づきチロシンキナーゼ間のクロストークに関する知見を得ることを目標として研究を進めます。
チロシンキナーゼは細胞内シグナル伝達のトリガーとなる酵素です。その活性は、活性化ループにあるチロシン残基のリン酸化により制御されます。受容体型チロシンキナーゼは、ホモ2量体形成を基軸としてキナーゼドメイン同士が近接し、互いにリン酸化し合うことで活性化されます。キナーゼによってはホモだけではなく、ヘテロで活性化されるものもあります。しかしながら、ヘテロの活性化に関する理解はホモの活性化に比べて進んでいません。さらには、細胞外領域を持たない非受容体型チロシンキナーゼについては、「どのチロシンキナーゼが」、「どのチロシンキナーゼを」活性化するかについての解明はほとんど進んでいないのが現状です。2006年にKuriyanらはEGFRのキナーゼドメイン間2量体構造を報告しました (Zhang et al., Cell, 2006)。それ以降、チロシンキナーゼがキナーゼドメイン間2量体構造を形成し、それが活性化において必須であることが明らかにされつつあります。そのため、このキナーゼドメイン間2量体構造の相互作用面をキナーゼ間で比較することにより、キナーゼ間クロストークを予測するための有用な知見が得られると考えられます。キナーゼドメイン間の相互作用は弱く、過渡的であり、解析は容易ではありません。これまでに、キナーゼドメイン間2量体構造は数例の報告があるのみであり、構造生物学的解析はほとんど進んでいません。活性化ループのリン酸化に関わるキナーゼドメイン間2量体構造に限定すると、IGF1R (Wu et al., EMBO J., 2008)とFGFR1 (Kobashigawa et al., Genes Cells, 2015)の2例のみとなります。キナーゼ間クロストークの予測に繋げるためには、より多くの種類のチロシンキナーゼについて活性化ループのリン酸化に関わる2量体構造情報を蓄積していくことが不可欠です。本研究課題では、その最初の段階として、FGFRファミリーの受容体型チロシンキナーゼに着目して研究を行います。
参考文献
- Kobashigawa Y, Amano S, Yoza K, Himeno R, Amemiya S, Morioka H, Yokogawa M, Kumeta H, Schlessinger J, Inagaki F. Nuclear magnetic resonance analysis of the conformational state of cancer mutant of fibroblast growth factor receptor 1 tyrosine kinase domain. Genes Cells. (2016) 21:350-357.
- Fukuda N, Suwa Y, Uchida M, Kobashigawa Y, Yokoyama H, *Morioka H. Role of the mobility of antigen binding site in high affinity antibody elucidated by surface plasmon resonance. J Biochem. (2017) 161:37-43.
- Yoza K, Himeno R, Amano S, Kobashigawa Y, Amemiya S, Fukuda N, Kumeta H, Morioka H and Inagaki F. Biophysical characterization of drug-resistant mutants of fibroblast growth factor receptor 1. Genes Cells. (2016) 21:350-357.
- Kobashigawa Y, Amano S, Yokogawa M, Kumeta H, Morioka H, Inouye M, Schlessinger J, Inagaki F. Structural analysis of the mechanism of phosphorylation of a critical autoregulatory tyrosine residue in FGFR1 kinase domain. Genes Cells. (2015) 20:860-870.
- Saio T, Ogura K, Kumeta H, Kobashigawa Y, Shimizu K, Yokochi M, Kodama K, Yamaguchi H, Tsujishita H, Inagaki F. Ligand-driven conformational change of MurD visualized by paramagnetic NMR. Scientific Reports. (2015) 5:e16685.
- Kobashigawa Y, Saio T, Ushio M, Sekiguchi M, Yokochi M, Ogura K, Inagaki F. Convenient method for resolving degeneracies due to symmetry of the magnetic susceptibility tensor and its application to pseudo contact shift-based protein-protein complex structure determination. J Biomol NMR. (2012) 53:53-63.
- Yokogawa M, Kobashigawa Y, Yoshida N, Ogura K, Harada K, Inagaki F. NMR analyses of the interaction between the FYVE domain of early endosome antigen 1 (EEA1) and phosphoinositide embedded in a lipid bilayer. J Biol Chem. (2012) 287:34936-45.
- Kobashigawa Y, Tomitaka A, Kumeta H, Noda NN, Yamaguchi M, Inagaki F. Autoinhibition and phosphorylation-induced activation mechanisms of human cancer and autoimmune disease-related E3 protein Cbl-b. Proc Natl Acad Sci USA. (2011) 108:20579-20584.
- Kobashigawa Y, Harada K, Yoshida N, Ogura K, Inagaki F. Phosphoinositide-incorporated lipid-protein nanodiscs: A tool for studying protein-lipid interactions. Anal Biochem. (2011) 410:77-83.








